Per integrazione virale si intende il fenomeno per cui un genoma virale si integra nel DNA della cellula ospite, in modo che quest’ultima non ne venga distrutta ma allorché si duplica lo trasmetta alle cellule figlie.
È un evento comune nella biologia di tutti gli Hepadnavirus ed ha luogo anche nel ciclo biologico del virus dell’epatite B umano, con importanti conseguenze cliniche.
L’integrazione dell’HBV-DNA correla con la presenza dell’epatocarcinoma (HCC); l’85-90% degli HCC associati all’epatite cronica B contengono sequenze virali integrate (1), molto meno frequenti nel tessuto epatico non tumorale. Il tumore epatico HBsAg-positivo presenta elevata instabilità genetica caratterizzata da rimodulazione cromosomica (2), tuttavia si ritiene che l’integrazione non sia capace di iniziare l’oncogenesi ma abbia un ruolo nella progressione del tumore.
Integrazione dell’HBV-DNA
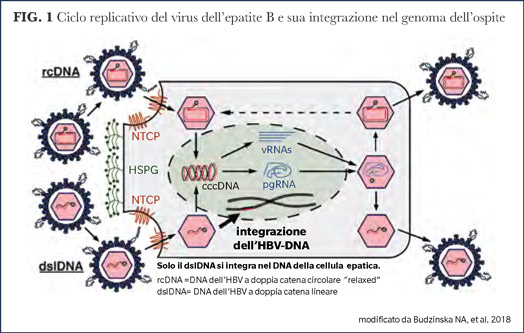 L’integrazione dell’HBV-DNA non è solo un fattore generale di oncogenesi ma può alterare l’equilibrio dell’espressione degli antigeni virali a favore dell’HBsAg. L’HBV maturo ed infettivo è costituito da un nucleocapside avvolto dall’HBsAg. Il nucleocapside contiene il DNA virale a doppia catena che esiste in due forme, come DNA a doppia catena circolare sciolta (relaxed, rcDNA) e, meno frequentemente, come DNA a doppia catena lineare (dslDNA) (Figura 1) (3). Trasportate nel nucleo degli epatociti, il loro destino diverge; RcDNA è convertito dagli enzimi cellulari nel covalently closed circular DNA dell’HBV (cccDNA), che regola la sintesi del virus, mentre dslDNA può divenire integrato nel genoma della cellula infetta, incorporandosi dove esistono fratture nel DNA della cellula (4).
L’integrazione dell’HBV-DNA non è solo un fattore generale di oncogenesi ma può alterare l’equilibrio dell’espressione degli antigeni virali a favore dell’HBsAg. L’HBV maturo ed infettivo è costituito da un nucleocapside avvolto dall’HBsAg. Il nucleocapside contiene il DNA virale a doppia catena che esiste in due forme, come DNA a doppia catena circolare sciolta (relaxed, rcDNA) e, meno frequentemente, come DNA a doppia catena lineare (dslDNA) (Figura 1) (3). Trasportate nel nucleo degli epatociti, il loro destino diverge; RcDNA è convertito dagli enzimi cellulari nel covalently closed circular DNA dell’HBV (cccDNA), che regola la sintesi del virus, mentre dslDNA può divenire integrato nel genoma della cellula infetta, incorporandosi dove esistono fratture nel DNA della cellula (4).
Mentre il cccDNA funziona da cervello genetico che codifica per tutte le proteine dell’HBV, compreso l’RNA pregenomico che verrà trascritto dalla polimerasi virale nel virus completo, il dslDNA non può generare nuovi virioni, in quanto incapace di codificare per l’RNA pregenomico; tuttavia, poiché il suo codice di lettura per l’HBsAg rimane funzionale, questo antigene può essere espresso dal DNA virale integrato anche quando il cccDNA è inattivato od eliminato (5). Ne deriva che nel corso dell’infezione da HBV, la sintesi dell’HBsAg può derivare da due fonti indipendenti, sia dalla trascrizione da parte del cccDNA che dalla trascrizione da parte del gene HBsAg integrato; l’HBsAg integrato può servire da mantello di rivestimento esterno (capside) nella morfogenesi del virione dell’HBV come quello trascritto dal cccDNA.
Fonte importante di HBsAg
L’espressione dell’HBsAg integrato non correla con la sintesi virale (cioè col titolo di HBV DNA nel siero); nei portatori di HBsAg HBeAg-negativi prevale rispetto a quello trascritto dal cccDNA nei portatori HBeAgpositivi. Studi trasversali hanno dimostrato che il rapporto fra HBsAg e HBV DNA è cento volte superiore nei pazienti HBeAg-negativi (6,7) ed uno studio longitudinale ha confermato che i livelli di HBsAg rimangono alti dopo l’eliminazione spontanea dell’HBeAg, malgrado riduzioni profonde dell’HBV DNA circolante (8). Parimenti, in uno studio recente di cinque espianti di fegato da pazienti con cirrosi da HBV e da HDV, i trascritti HbsAg integrati prevalevano su quelli derivati dal cccDNA, a dimostrazione che il genoma virale integrato è una sorgente importante di HBsAg anche nelle epatopatie avanzate (9).
L’espressione alternativa dell’HBsAg dall’HBV integrato nel genoma dell’ospite ha importanti conseguenze cliniche.
lL’HBsAg è presente in circolo in gran quantità, fino a 1012/ml di particelle non-infettive vuote di DNA virale. L’HBsAg in eccesso nel sangue contribuisce all’esaurimento della risposta immunologica da parte dell’organismo infetto (10); agisce come esca che assorbe gli anticorpi neutralizzanti il virus, ed il suo eccesso contribuisce dunque alla soppressione delle risposte anticorpali. Si assume che la riduzione dei livelli di HBsAg contribuisca a recuperare la risposta immune permettendo la clearance degli epatociti infetti.
lL’accumulo di HBsAg negli epatociti è considerato un fattore di rischio specifico per l’insorgenza dell’HCC indipendentemente dall’entità della replicazione virale; l’accumulo della proteina preS1 large dell’antigene e di quelle preS2/S mutanti inducono modificazioni che contribuiscono alla trasformazione tumorale degli epatociti (11).
lSebbene l’HBsAg sia considerato un marcatore di attività dell’HBV e la sua diminuzione quantitativa è usata per valutate l’efficacia della terapia con il Peg-IFN o gli analoghi nucleosidici contro l’HBV, per la sua origine alternativa da DNA integrato non suscettibile alle terapie antivirali, esso rappresenta un marcatore inaffidabile di risposta ai farmaci antivirali convenzionali.
lLe attuali terapie contro l’HBV sono dirette ad inibire la replicazione virale, diminuiscono l’HBsAg trascritto dal cccDNA ma non hanno efficacia sull’HBsAg integrato (12); quest’ultima è l’unica risorsa dell’HBV necessaria al virus dell’epatite D per assemblarsi nel virione infettivo e suscitare l’epatite D; finché non viene eliminato la malattia può recidivare (13).
Sviluppo di nuovi farmaci
Non sorprende dunque che i farmacologi e l’industria farmaceutica siano da tempo impegnati nello sviluppo di nuovi farmaci mirati ad eliminare l’HBsAg di qualunque origine, complementari ai validi antivirali nucleosidici che controllano la replicazione virale e aboliscono l’infiammazione epatica. I più probabili candidati sono gli RNA interferenti specifici (siRNA) capaci di esautorare tutti i trascritti virali sia quelli codificati dal cccDNA che quelli codificati dal genoma integrato (14); uno degli siRNA più promettenti è VIR-2218; disegnato per silenziare i trascritti di tutti i dieci genotipi dell’HBV, in un recente trial di fase 2 ha dimostrato buona tolleranza e iniziale efficacia (15).
Bibliografia
- Mason WS, Liu C, Aldrich CE, et al. Clonal expansion of normal-appearing human hepatocytes during chronic hepatitis B virus infection. J. Virol. 2010; 84:8308-8315.
- Tu T, Mason WS, Clouston AD, et al. Clonal expansion of hepatocytes with a selective advantage occurs during all stages of chronic hepatitis B virus infection. J. Viral Hepat. 2015; 22: 737-753.
- Budzinska MA, Shackel NA, Urban S, et al. Cellular genomic sites of hepatitis B virus DNA integration. Genes 2018; 9: 365.
- Bill CA, Summers J. Genomic DNA double-strand breaks are targets for hepadnaviral DNA integration. Proc. Natl. Acad. Sci. USA 2004; 101: 11135-11140.
- Freitas N, Cunha C, Menne S, Gudima SO. Envelope proteins derived from naturally integrated hepatitis B virus DNA support assembly and release of infectious hepatitis Delta virus particles. J. Virol. 2014; 8: 5742-5754.
- Larsson SB, Eilard A, Malmstrom S, et al. HBsAg quantification for identification of liver disease in chronic hepatitis B virus carriers. Liver Int 2014; 34:e238-e245.
- Larsson SB, Malmstrom S, Hannoun C, et al. Mechanisms downstream of reverse transcription reduce serum levels of HBV DNA but not of HBsAg in chronic hepatitis B virus infection. Virology J 2015; 12: 213.
- Tripodi G, Larsson SB, Norkrans G, Lindh M. Smaller reduction of hepatitis B virus DNA in liver tissue than in serum in patients losing HBeAg. J Med Virol 2017; 89: 1937-1943.
- Ringlander J, Skoglund C, Prakash K, et al. Deep sequencing of liver explant transcriptomes reveals extensive expression from integrated hepatitis B virus DNA. J Viral Hep 2020; 27: 1162-1170.
- Bertoletti A, Ferrari C. Adaptive immunity in HBV infection. J Hepatol 2016; 64: S71-S83.
- Levrero M, Zucman-Rossi J. Mechanisms of HBV-induced hepatocellular carcinoma. Hepatol 2016; 64 (Suppl. 1): S84-s101.
- Lampertico P, Agarwal K, Berg T, et al. EASL 2017 Clinical practice guidelines on the management of hepatitis B virus infection. J Hepatol 2017; 67: 370-398.
- Caviglia GP, Rizzetto M. Treatment of hepatitis D: an unmet medical need. Clin Microbiol Infect. 2020; 26: 824-827.
- Wooddell CI, Yuen MF, Chan HL, et al RNAi-based treatment of chronically infected patients and chimpanzees implicates integrated hepatitis B virus DNA as a source of HBsAg. Sci Transl Med. 2017; 9(409): eaan0241.
- Gane E, Lim Y-S, Tangkijvanich P, et al. Preliminary safety and antiviral activity of VIR-2218, an X- targeting HBV RNAi therapeutic, in chronic hepatitis B patients. EASL 2020 Abs AS068 J Hepatol 2020; 73 (Suppl1): S50.






