Nonostante il successo della terapia antiretrovirale (ART) nel garantire il raggiungimento e il mantenimento della soppressione virologica, la valutazione della farmacoresistenza rimane ancora un fattore cruciale da prendere in considerazione per la prevenzione e la gestione del fallimento della terapia sia nel paziente naive alla ART ma anche nei pazienti in trattamento (sia in fallimento che in soppressione virologica) (1,2). Il test di resistenza genotipica effettuato mediante il classico sequenziamento Sanger sull'HIV-RNA plasmatico ha supportato efficacemente la ART per lungo tempo. Oggi sono disponibili test di genotipizzazione ancora non standardizzati per la routine clinica il cui utilizzo può garantire l’efficacia terapeutica a lungo termine: il test su cellule mononucleate da sangue periferico (PBMC) e il test eseguito mediante sequenziamento massivo parallelo (ossia Next Generation Sequencing, NGS).
Valutazione della farmacoresistenza nei pazienti con bassi livelli di viremia
Nei pazienti sotto controllo virologico o con viremia rilevabile ma bassa (<1000 copie/ml), il test di resistenza genotipica eseguito su PBMC è tecnicamente fattibile e può rappresentare un valido strumento per definire profili di farmacoresistenza archiviati nel DNA provirale.
Tuttavia, i risultati ottenuti devono essere interpretati con cautela in quanto il genotipo su PBMC potrebbe non rilevare precedenti mutazioni di resistenza, o al contrario, potrebbe rilevare mutazioni clinicamente irrilevanti. Pertanto, la genotipizzazione del DNA provirale di routine è attualmente suggerita dalle linee guida, ma non raccomandata (1,2). Nonostante ciò, ove sia disponibile, il test su DNA provirale può fornire informazioni utili. Ad esempio, nei pazienti virologicamente soppressi per i quali viene pianificato un cambio terapeutico, la genotipizzazione su DNA provirale può essere utile in persone con molteplici fallimenti virologici e/o con storia di resistenza non disponibile. Uno studio ha messo in evidenza che in pazienti virologicamente soppressi per i quali era stato effettuato un test genotipico su PBMC prima del cambio terapeutico, coloro che avevano un virus con resistenza intermedia o totale avevano una maggiore probabilità di avere un rialzo viremico rispetto a quelli con un virus completamente suscettibile. Valutando la resistenza sia nei PBMC che nel genotipo storico da plasma, la più alta probabilità di rialzo viremico era nei pazienti per i quali la resistenza era stata rilevata in entrambi i compartimenti (3) (Figura 1).
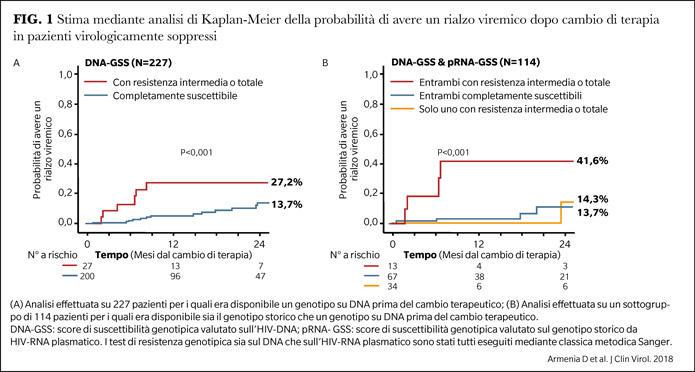
Questi risultati supportano l'ipotesi che la resistenza rilevata nei PBMC sia clinicamente rilevante non solo per i pazienti con scarse informazioni precedenti, ma anche per quelli con una storia di resistenza completa (2).
Valutazione della farmacoresistenza: dal Sanger all’NGS
Nonostante l’esteso utilizzo del test di resistenza genotipica mediante il classico sequenziamento Sanger, le informazioni sulla resistenza virale che esso fornisce rappresentano solo la punta dell’iceberg. Infatti con tale approccio è possibile rilevare solo le varianti virali resistenti maggioritarie, presenti nell’ospite con una prevalenza maggiore del 15-20% (4).
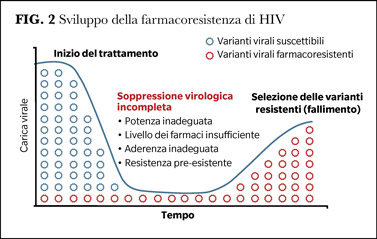 L’introduzione dell’NGS ha permesso di superare questo limite. Infatti, mediante l’NGS è oggi possibile valutare la variabilità del genoma di HIV all’interno dell’ospite e individuare mutazioni di resistenza presenti anche a bassa prevalenza fino allo 0,1% (5). L’identificazione di queste varianti resistenti minoritarie è importante perché, in condizioni di incompleta soppressione virologica, possono determinare l’emergenza di resistenza e, di conseguenza, il fallimento della ART (Figura 2).
L’introduzione dell’NGS ha permesso di superare questo limite. Infatti, mediante l’NGS è oggi possibile valutare la variabilità del genoma di HIV all’interno dell’ospite e individuare mutazioni di resistenza presenti anche a bassa prevalenza fino allo 0,1% (5). L’identificazione di queste varianti resistenti minoritarie è importante perché, in condizioni di incompleta soppressione virologica, possono determinare l’emergenza di resistenza e, di conseguenza, il fallimento della ART (Figura 2).
L'uso dell’NGS in individui con infezione da HIV di nuova diagnosi ha permesso di rilevare un'ampia percentuale di varianti di resistenza minoritarie che non sarebbero state rilevate mediante il tradizionale sequenziamento Sanger, incluse varianti di resistenza agli inibitori dell’integrasi (6,7). Questo risultato è degno di nota; infatti ad oggi la resistenza trasmessa a tale classe di farmaci mediante approccio Sanger è risultata molto rara. Pertanto, sono importanti studi di sorveglianza globale mediante NGS al fine di fornire una valutazione più completa della prevalenza della resistenza trasmessa (inclusa quella agli inibitori dell’integrasi) nelle diverse aree geografiche (6,7).
In individui ART-experienced, lo score di suscettibilità genotipica (utilizzato per valutare l’attività dei farmaci antiretrovirali) calcolato mediante NGS è risultato più sensibile nell’avere un valore predittivo sull’outcome virologico sotto regimi di salvataggio rispetto a quello valutato mediante metodica Sanger (8).
In individui virologicamente soppressi, il rilevamento della farmacoresistenza su DNA provirale è più accurato se effettuato mediante NGS piuttosto che tramite il classico sequenziamento Sanger.
Un aspetto importante ancora da definire nella valutazione della presenza delle varianti minoritarie di resistenza mediante NGS è quale sia la soglia clinicamente rilevante da utilizzare. Essa dipende da diversi fattori: la classe di farmaci antiretrovirali, il tipo di farmaco all’interno della stessa classe (in relazione ad esempio alla sua barriera genetica), le specifiche mutazioni, il carico virale (altresì chiamato mutational load).
Riguardo la classe dei farmaci, diversi studi in passato hanno dimostrato come la presenza di varianti minoritarie associate a resistenza agli inibitori non nucleosidici della trascrittasi inversa (NNRTI) sia associata ad un maggiore rischio di fallimento virologico in individui che iniziavano un primo regime terapeutico con tale classe di farmaci (4). Tali studi hanno messo in evidenza anche come la quantità di tali varianti minoritarie, piuttosto che la loro mera presenza, sia un fattore importante nel regolare l’efficacia dei primi regimi contenenti NNRTI. è pertanto importante considerare il mutational load, ossia la frequenza delle specifiche varianti minoritarie in relazione alla carica virale o ai livelli di HIV-DNA (4,8). Non sono ad oggi disponibili dati sull’impatto delle varianti minoritarie sull’outcome virologico durante i nuovi regimi includenti ad esempio farmaci ad alta barriera genetica come gli inibitori della proteasi potenziati o gli inibitori dell’integrasi di seconda generazione.
Valutazione delle mutazioni inusuali sulle sequenze di DNA provirale
In individui in trattamento antiretrovirale, un importante aspetto da prendere in considerazione nella valutazione della farmacoresistenza su PBMC è la ridotta specificità del test genotipico su tale compartimento dovuta ad un'alta percentuale di provirus difettivi o silenziati episomicamente. Infatti, la caratterizzazione genetica dei provirus di HIV-1 isolati da cellule T CD4+ di individui in ART efficace ha rivelato che solo il 2-12% del virus persistente è geneticamente intatto e potenzialmente competente per la replicazione (9). Il resto dei genomi integrati di HIV presenta importanti anomalie della sequenza che sono incompatibili con la replicazione virale, comprese grandi delezioni, ed ipermutazioni G->A mediate dal polipeptide catalitico (APOBEC) che di solito provocano mutazioni letali, quali codoni di stop e mutazioni in posizioni altamente conservate.
è stato osservato che nelle sequenze provirali intatte il tasso di mutazioni associate a resistenza è pressoché nullo, mentre aumenta significativamente (intorno allo 0,25) nelle sequenze difettive. Nello specifico, nelle sequenze difettive la presenza di ipermutazioni è associata ad un tasso più elevato di mutazioni di resistenza, portando ad una potenziale sovrastima della presenza di tali mutazioni (10).
Nelle sequenze di DNA provirale ottenute mediante NGS, la presenza di ipermutazioni, così come di altre mutazioni inusuali dovute ad artefatti di sequenza, aumenta per soglie di rilevamento <5% (5).
Pertanto, è necessaria un’attenta valutazione della presenza di queste mutazioni a soglie di rilevamento basse; inoltre la soglia specifica alla quale viene rilevata ciascuna mutazione di resistenza deve essere inclusa nel report del genotipo.
Messaggi-chiave
- Oggi, nonostante il miglioramento della terapia antiretrovirale, il test di resistenza genotipica è ancora cruciale per la prevenzione e la gestione del fallimento della terapia.
- Test molecolari più altamente sensibili e specifici sono fondamentali per il monitoraggio a lungo termine dell'infezione da HIV.
- Il miglioramento dei test di resistenza (grazie all'utilizzo del test su HIV-DNA e dell’NGS) è finalizzato a rilevare mutazioni di resistenza a bassa prevalenza anche a viremia non rilevabile.
- L’NGS è estremamente più sensibile del classico sequenziamento mediante metodica Sanger e fornisce ulteriori informazioni potenzialmente utili sulla presenza delle mutazioni di resistenza.
- Tuttavia, sono necessarie ulteriori indagini per definire le soglie clinicamente rilevanti per predire il fallimento virologico.
- European AIDS Clinical Society (EACS), European AIDS Clinical Society (EACS) Guidelines October 2021. Version 11.0. 2021. Disponibile su: https://www.eacsociety.org/media/final2021eacsguidelinesv11.0_oct2021.pdf
- DHHS Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the Use of Antiretroviral Agents in Adults and Adolescents Liv- ing with HIV Developed by the DHHS Panel on Antiretroviral Guide- lines for Adults 2022. Disponibile su: https://clinicalinfo.hiv.gov/sites/default/files/https://clinicalinfo.hiv.gov/sites/default/files/guidelines/documents/AdultandAdolescentGL.pdf
- Armenia D, Zaccarelli M, Borghi V, et al. Resistance detected in PBMCs predicts virological rebound in HIV-1 suppressed patients switching treatment. J Clin Virol. 2018;104:61-64.
- Chabria SB, Gupta S, Kozal MJ. Deep sequencing of HIV: clinical and research applications. Annu Rev Genomics Hum Genet. 2014; 15:295-325.
- Tzou PL, Kosakovsky Pond SL, et al. Analysis of unusual and signature APOBEC-mutations in HIV-1 pol next-generation sequences. PLoS One. 2020; 15(2):e0225352.
- Baxter JD, Dunn D, Tostevin A, et al. Transmitted HIV-1 drug resistance in a large international cohort using next-generation sequencing: results from the Strategic Timing of Antiretroviral Treatment (START) study. HIV Med. 2021; 22:360-371.
- Mbisa JL, Ledesma J, Kirwan P, et al. Surveillance of HIV-1 transmitted integrase strand transfer inhibitor resistance in the UK. J Antimicrob Chemother. 2020; 75:3311-3318.
- Armenia D, Santoro MM, Bellocchi MC, et al. PRESTIGIO Registry Study Group. Viral resistance burden and APOBEC editing correlate with virological response in heavily treatment-experienced people living with multi-drug resistant HIV. Int J Antimicrob Agents. 2022; 59(1):106492.
- Palmer S. The HIV-1 proviral landscape: what have we learned? 29th Conference on retroviruses and opportunistic infections (CROI); 12-16 February 2022. Abstract 107.
- Li Y, Etemad B, Dele-Oni R, et al. Drug resistance mutations in HIV provirus are associated with defective proviral genomes with hypermutation. AIDS. 2021; 35:1015-1020.






