La diffusione del lignaggio Omicron a fine 2021 e la selezione continua di sottovarianti con nuove mutazioni nella Spike, anche localizzate nel dominio di legame con il recettore (RBD), ha determinato la necessità di rivalutare gli approcci preventivi e terapeutici nei confronti del SARS-CoV-2. Numerosi sono i lavori che hanno indagato la capacità infettiva e la suscettibilità delle nuove varianti ai vaccini mono/bivalenti e agli anticorpi monoclonali (mAbs), concentrandosi prevalentemente sulla capacità neutralizzante di entrambi e non sempre con risultati paragonabili a causa della mancata standardizzazione dei protocolli sperimentali utilizzati. Inoltre, l’efficacia di un vaccino o di un mAb viene conferita da una molteplicità di fattori, che concorrono alla limitazione del danno e della replicazione virale.
Addetia e coll, con un lavoro pubblicato su Nature nel settembre del 2023 (1), indagano la complessità dei fattori che concorrono al successo delle strategie vaccinali e terapeutiche, attraverso la caratterizzazione puntuale delle varianti Omicron prevalenti tra la fine del 2022 e l’inizio del 2023 (es. BQ.1.1, BA.2.75.2 e le forme ricombinanti XBB.1, XBB.1.5) sotto il profilo della:
- capacità infettiva
- suscettibilità ai diversi mAbs
- risposta al vaccino mono/bivalente ad mRNA.
La capacità infettiva è stata stimata attraverso l’affinità di legame tra il dominio extracellulare di ACE2 e l’RBD e la capacità fusogena. In primo luogo, la costante di dissociazione tra ACE2 ed RBD, misura dell’affinità di legame, è risultata simile per le varianti BQ.1.1, BA.2.75.2 e XBB.1.5 e del tutto paragonabile alla BA.5. Pur utilizzando una entrata endosoma-mediata, indipendente dal TMPRSS2, tali varianti promuovono però una fusione più efficiente tra il pericapside virale e la membrana endosomale, comportando una migliore cinetica di replicazione virale, come già riscontrato nel clade Delta (2). Ciò spiegherebbe dunque la loro rapida diffusione a discapito della stessa BA.5.
 Guardando alla struttura della Spike, alcune mutazioni selezionate in tali varianti, come la K444T nella BQ.1.1, la R446T nelle BQ.1.1 e XBB.1 e la G446S+V445P nella XBB.1 causano una pressocché totale inibizione di legame tra una vasta gamma di mAbs e gli epitopi target, confermando i dati di neutralizzazione in vitro (3). Diverso è il caso di sotrovimab, che strutturalmente dimostra di mantenere il legame tra il frammento legante l’antigene (Fab) ed il suo epitopo, pur perdendo alcuni gradi di affinità rispetto al ceppo originale di Wuhan, confermando anche in questo caso molti dei dati sulla capacità di neutralizzazione di tale mAb sino ad ora pubblicati (4-5). Tuttavia, l’efficienza di sotrovimab nell’inibire la quasi totalità delle varianti Omicron selezionate nel tempo risiede in vari aspetti che caratterizzano tale mAb, e che non sono solo relativi alla conservazione dell’epitopo target, interno all’RBD ma non coinvolto nel sito di legame ad ACE2 (6), ma anche alla lunga emivita, ed il mantenimento di una buona funzione effettrice del frammento cristallizzabile (Fc) dell’anticorpo (7). A questo proposito, Addetia e coll. hanno indagato la capacità di sotrovimab di attivare una citotossicità e fagocitosi anticorpo-dipendente, caratteristica non comune alla maggior parte dei mAbs disponibili contro SARS-CoV-2.
Guardando alla struttura della Spike, alcune mutazioni selezionate in tali varianti, come la K444T nella BQ.1.1, la R446T nelle BQ.1.1 e XBB.1 e la G446S+V445P nella XBB.1 causano una pressocché totale inibizione di legame tra una vasta gamma di mAbs e gli epitopi target, confermando i dati di neutralizzazione in vitro (3). Diverso è il caso di sotrovimab, che strutturalmente dimostra di mantenere il legame tra il frammento legante l’antigene (Fab) ed il suo epitopo, pur perdendo alcuni gradi di affinità rispetto al ceppo originale di Wuhan, confermando anche in questo caso molti dei dati sulla capacità di neutralizzazione di tale mAb sino ad ora pubblicati (4-5). Tuttavia, l’efficienza di sotrovimab nell’inibire la quasi totalità delle varianti Omicron selezionate nel tempo risiede in vari aspetti che caratterizzano tale mAb, e che non sono solo relativi alla conservazione dell’epitopo target, interno all’RBD ma non coinvolto nel sito di legame ad ACE2 (6), ma anche alla lunga emivita, ed il mantenimento di una buona funzione effettrice del frammento cristallizzabile (Fc) dell’anticorpo (7). A questo proposito, Addetia e coll. hanno indagato la capacità di sotrovimab di attivare una citotossicità e fagocitosi anticorpo-dipendente, caratteristica non comune alla maggior parte dei mAbs disponibili contro SARS-CoV-2.
Sebbene infatti il potere neutralizzante di sotrovimab risenta dell’affinità di legame tra il Fab e l’epitopo target, e dunque registri una riduzione dell’attività neutralizzante per alcune varianti, la magnitudo dell’attività citotossica e fagocitica risulta invariata in tutte le varianti analizzate. Tale evidenza ottenuta in vitro è stata inoltre confermata nel modello animale, in cui la somministrazione preventiva di sotrovimab è stata in grado di ridurre la perdita di peso ed abbattere sensibilmente la carica virale nel polmone dopo infezione con BQ.1.1 e XBB.1.5 (Figura 1).
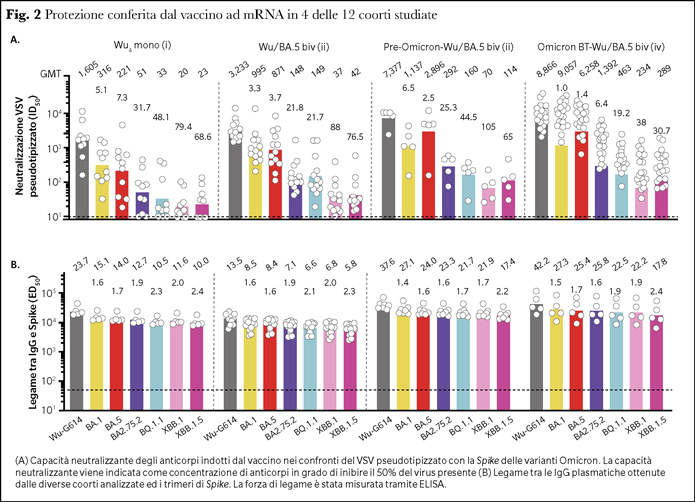
L’ultima parte del lavoro di Addetia e coll. ispeziona l’efficacia del vaccino anti-SARS-CoV-2 a mRNA nei confronti delle già citate varianti Omicron. Per tale scopo, sono stati collezionati sieri da un vasto numero di coorti, permettendo di rappresentare al meglio il contesto epidemiologico globale del periodo in cui il lavoro è stato concepito e scritto. Le coorti spaziavano da soggetti vaccinati 4 volte con l’mRNA della Spike di Wuhan (monovalente) e mai infettati, a soggetti a cui erano stati somministrati sia vaccini monovalenti sia bivalenti composti da Wuhan/BA.5. I risultati ottenuti confermano la maggiore attività neutralizzante delle vaccinazioni bivalenti rispetto al vaccino monovalente nei confronti delle varianti XBB.1, XBB.1.5, BA.2.75.2 e BQ.1.1, indipendentemente da una precedente o meno infezione (Figura 2A). Oltre agli anticorpi neutralizzanti generati dalla somministrazione vaccinale, la totalità delle IgG prodotte rimane in grado di legare l’RBD o i trimeri di Spike indipendentemente dalle varianti Omicron, promuovendo la lisi delle cellule infette da parte delle cellule citotossiche (Figura 2B), e dunque contribuendo alla protezione dalla malattia severa. Infine, le cellule B della memoria completano il pathway cellulare che coopera nella risposta vaccinale. Gli autori dimostrano come l’imprinting immunitario sia fondamentale per richiamare in corso di infezione anticorpi diretti nei confronti della proteina Spike utilizzata nelle prime dosi vaccinali e sottolineano come una prima vaccinazione bivalente Wuhan/BA.5 permetta un arricchimento di cellule B della memoria reattive anche nei confronti di nuove varianti.
Riportando l’attenzione all’attuale situazione epidemiologica italiana ed europea, è necessario sottolineare la larga diffusione delle varianti EG.5.1 e XBB.2.3, discendenti delle forme ricombinanti XBB.1.9 e XBB.2 ed emerse successivamente al lavoro di Addetia e coll. (1) Le mutazioni nell’RBD caratterizzanti tali varianti (la F456L per la EG.5.1 e la S486P per la XBB.2.3) non pregiudicano tuttavia l’efficacia di sotrovimab né degli anticorpi generati grazie al vaccino bivalente (8-9), mantenendo così invariato il valore scientifico di tali considerazioni.
- Addetia A, Piccoli L, Case JB, et al. Neutralization, effector function and immune imprinting of Omicron variants. Nature. 2023;621(7979):592-601.
- Saito A, Irie T, Suzuki R, et al. Enhanced fusogenicity and pathogenicity of SARS-CoV-2 Delta P681R mutation. Nature. 2022;602(7896):300-306.
- Wang Q, Iketani S, Li Z, et al. Alarming antibody evasion properties of rising SARS-CoV-2 BQ and XBB subvariants. Cell. 2023;186(2):279-286.e8.
- Arora P, Cossmann A, Schulz SR, et al. Neutralisation sensitivity of the SARS-CoV-2 XBB.1 lineage. Lancet Infect Dis. 2023;23(2):147-148.
- Touret F, Giraud E, Bourret J, et al. Enhanced neutralization escape to therapeutic monoclonal antibodies by SARS-CoV-2 omicron sub-lineages. iScience. 2023;26(4):106413.
- Pinto D, Park YJ, Beltramello M,et al. Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature. 2020;583(7815):290-295.
- Case JB, Mackin S, Errico JM, et al. Diamond MS. Resilience of S309 and AZD7442 monoclonal antibody treatments against infection by SARS-CoV-2 Omicron lineage strains. Nat Commun. 2022;13(1):3824.
- Julia N. Faraone, Panke Qu, et al. Immune evasion and membrane fusion of SARS-CoV-2 XBB subvariants EG.5.1 and XBB.2.3, Emerging Microbes & Infections. 2023;12:2.
- Zhang L, Kempf A, Nehlmeier I, et al. Neutralisation sensitivity of SARS-CoV-2 lineages EG.5.1 and XBB.2.3. Lancet Infect Dis. 2023;23(10):e391-e392.






