Dagli inizi di gennaio 2020, l’attenzione della comunità medico-scientifica e degli organi di divulgazione è focalizzata sull’epidemia causata dal nuovo coronavirus (SARSCoV- 2) emerso a Wuhan in Cina a fine 2019 e che si sta ancora diffondendo soprattutto in una vasta regione dall’epicentro (con più di 80.000 casi a fine febbraio 2020) nonostante gli sforzi senza precedenti messi in atto dalla Cina per contenerlo e che hanno determinato una significativa diminuzione dei casi nell’ultima settimana di febbraio. L’infezione causata dal nuovo coronavirus (definita Covid-19, ovvero: “coronavirus disease-2019”) colpisce l’apparato respiratorio con un ampio spettro di sintomatologia: da lieve a molto grave ad elevata mortalità a causa della polmonite virale per sindrome da distress respiratorio acuto (in inglese, ARDS) (1).
Precedentemente all’epidemia in corso di SARS-CoV-2, emersero due nuovi coronavirus che causarono le sindromi note come SARS (Severe Acute Respiratory Syndrome), scoppiata in Cina nel 2002 (2), e MERS (Middle East Respiratory Syndrome) emersa in Arabia Saudita nel 2012 (3). Mentre l’epidemia di SARS fu contenuta entro il mese di luglio del 2003, la MERS non è mai stata definitivamente eliminata in Medio Oriente.
La caratteristica comune di queste tre sindromi cliniche causate da coronavirus è di essere zoonosi, patologie umane causate da virus che normalmente circolano negli animali. Le zoonosi implicano una catena di trasmissione dal serbatoio naturale di infezione (per questi coronavirus i pipistrelli) ad animali che fungono da ospiti intermedi e amplificatori della replicazione virale (principalmente lo zibetto per SARS-CoV e i camelidi dell’area geografica per MERS-CoV, rispettivamente, mentre per il SARS-CoV-2 non è ancora stato identificato un ospite intermedio) prima di essere trasmessi all’uomo (4).
Il passaggio successivo è l’acquisizione della capacità di trasmissione interumana indipendente dalla fonte animale, fortunatamente molto limitata nel caso della MERS, ma purtroppo efficiente per le epidemie di SARS e ora per Covid-19.
Fattori che favoriscono la diffusione e persistenza dei coronavirus
La trasmissione zoonotica è una caratteristica peculiare dei coronavirus favorita dalla continua espansione della popolazione umana e dalla convivenza dell’uomo con animali domestici che, a loro volta, vengono in contatto con animali selvatici, soprattutto con i pipistrelli, principali serbatoi naturali dei coronavirus come di altre specie virali (5). In particolare, nel caso dei coronavirus, il salto di specie dall’animale all’uomo è favorito dalla loro intrinseca capacità di adattarsi rapidamente all’ospite infettato in seguito alla comparsa di mutazioni nel genoma virale a RNA, che insorgono spontaneamente durante la replicazione virale e sono favorite dalla pressione selettiva esercitata dalla risposta immunitaria.
Particolare importanza rivestono le mutazioni della proteina spike, presente sul mantello delle particelle virali (virioni), responsabile dell’ingresso del virus nella cellula attraverso il legame ad un recettore specifico mediato da un Receptor Binding Domain (RBD). Nel caso del SARS-CoV il recettore specifico è stato identificato nell’enzima Angiotensin Converting Enzyme 2 (ACE2) presente sulle cellule epiteliali dell’apparato respiratorio umano (6). E’ interessante sottolineare come negli ultimi mesi della trasmissione del SARS-CoV da uomo a uomo siano emerse mutazioni che ne hanno diminuito l’affinità di legame al recettore ACE2 umano (7) e potrebbero aver quindi contribuito alla limitazione della diffusione dell’infezione principalmente ottenuta grazie alle misure di contenimento (Figura 1A).
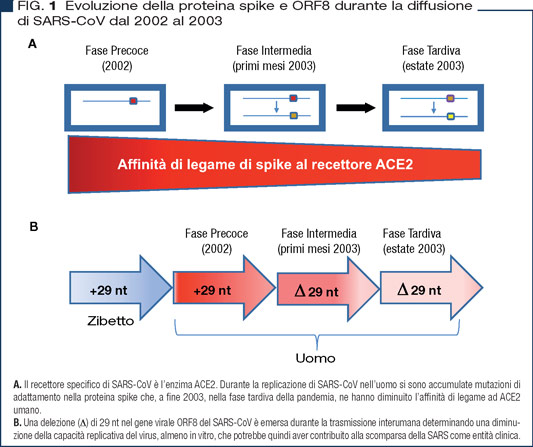
Inoltre, di particolare rilievo è stata l’emergenza di una delezione di 29 nucleotidi (nt) in un gene virale definito Open Reading Frame 8 (ORF8) del SARS-CoV. Questo gene ha, infatti, subito una graduale eliminazione durante l’epidemia umana in quanto i primi casi di infezione da SARS-CoV furono causati da un virus con un ORF8 intatto, come osservato anche nella maggioranza dei virus sequenziati nei pipistrelli e nello zibetto (Figura 1B). Studi di mutagenesi del SARS-CoV hanno dimostrato che questa delezione di 29 nt in ORF8 determina una diminuzione della capacità replicativa del virus, almeno in vitro (8), che potrebbe aver contribuito al contenimento della diffusione del virus.
I coronavirus hanno una capacità intrinseca di compiere salti di specie e adattarsi all’ospite mediante l’acquisizione di mutazioni che possono anche diminuirne l’infettività e alterarne la patogenicità. Sarà necessario prestare particolare attenzione al crogiolo di virus simili a SARS-CoV e SARS-CoV-2 presenti nei pipistrelli della caverna di Yunnan (9), e probabilmente altrove, in grado di legarsi efficientemente al recettore ACE2 umano per poter prevenire o, quantomeno, limitare prontamente future epidemie da coronavirus patogeni per l’uomo.
Bibliografia
- Li Q, Guan X, Wu P, et al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia. N Engl J Med 2020 doi:10.1056/NEJMoa2001316.
- Peiris JS, Guan Y, Yuen KY. Severe acute respiratory syndrome. Nat Med 2004; 10:S88-97.
- Zaki AM, van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia. N Engl J Med 2012; 367:1814-20.
- Cui J, Li F, Shi ZL. Origin and evolution of pathogenic coronaviruses. Nat Rev Microbiol 2019; 17:181-192.
- Graham RL, Donaldson EF, Baric RS. A decade after SARS: strategies for controlling emerging coronaviruses. Nat Rev Microbiol 2013; 11:836-48.
- Menachery VD, Yount BL, Jr., Debbink K, et.al. A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. Nat Med 2015; 21:1508-13.
- Li W, Zhang C, Sui J, et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2. EMBO J 2005; 24:1634-43.
- Muth D, Corman VM, Roth H, et al. Attenuation of replication by a 29 nucleotide deletion in SARS-coronavirus acquired during the early stages of human-to-human transmission. Sci Rep 2018; 8:15177.
- Hu B, Zeng LP, Yang XL, et al. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. PLoS Pathog 2017; 13:e1006698.






